宮崎大學終身教職推進室的福永圭佑副教授、九州大學研究生院農學研究院的寺本嶽大助教、角田佳充教授、沖繩科學技術大學院大學(OIST)核酸化學與工程單元的橫林洋平教授、東京科學大學地球生命研究所的松浦友亮教授等宣佈,成功運用X射線晶體結構分析方法,闡明瞭所開發的兩種人工RNA-蛋白質複合體(RNP)在結合模式上的差異。研究團隊利用這兩種RNP成功開發出了高性能核糖開關(用於基因表現調控的RNA開關),並在無細胞蛋白質合成系統(cell-free系統)中驗證了其優異性。相關研究成果已發表在國際學術期刊《Nucleic Acids Research》的3月23日刊上。
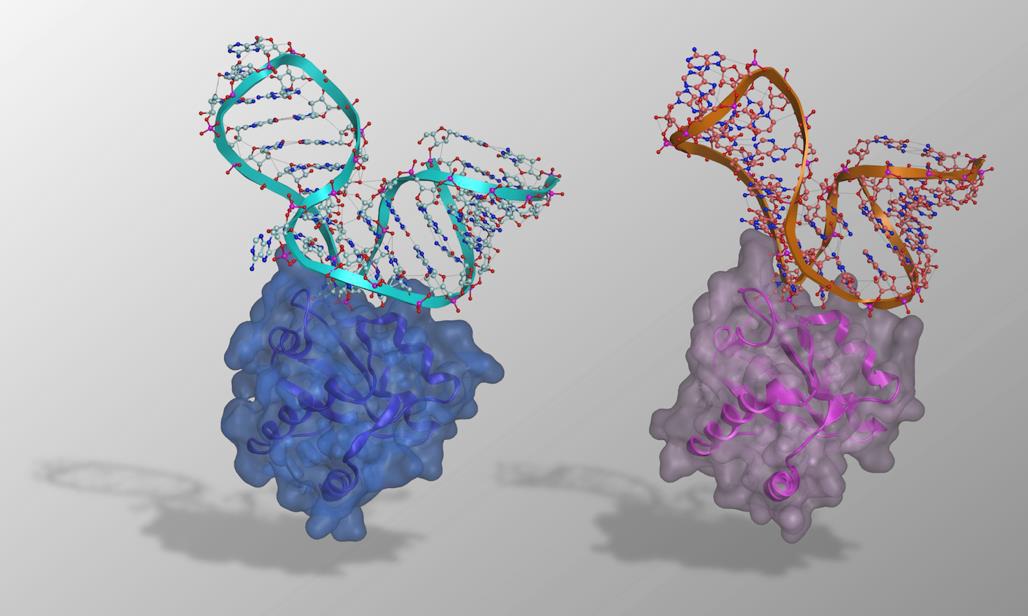
圖1. CS1-LS4(左)、CS2-LS12(右)的結構示意圖(供圖:宮崎大學)
無細胞蛋白質合成系統作為以醫療用途等為目標的特定蛋白質大量合成,以及作為生命化學研究的工具,相關研究正在全球範圍內推進。
mRNA結合蛋白在mRNA由細胞核內的DNA轉錄而來,並在核糖體翻譯的過程中發揮著核心作用。
此前,OIST的研究團隊開發出了能夠改變RNA-RBP間結合模式的PD-SELEX法。通過改造合成生物學研究中通常用作分子元件的古細菌來源L7Ae蛋白及其所結合的kink-turn RNA的結合選擇性,成功獲得了兩組正交性RNA-RBP配對——CS1-LS4與CS2-LS12。另一方面,這些高同源性的RNA-RBP配對能夠形成特異性配對的機制此前尚不明確。
對此,研究團隊採用X射線共晶結構分析技術,嘗試明確其相輔作用機制。
研究團隊成功確定了CS1-LS4和CS2-LS12的結構。並發現了這兩組RNP的整體結構與作為其祖先的kink-turn RNA-L7Ae複合體高度相似。
此外,研究團隊還探討了在無細胞轉錄翻譯系統中發揮作用的蛋白質配體響應型核糖開關的開發。
在T7啟動子序列後立即嵌入RNA適配體序列(CS1或CS2),設計出一種在無蛋白質配體(LS4或LS12)條件下通過遮蔽核糖體識別序列以抑制下游基因翻譯的機制,製作了僅在配體存在條件下進行蛋白質合成的ON開關。
為驗證其性能,研究團隊以螢光蛋白(EGFP)報告基因的表達水平為指標開展篩檢,確認該無細胞核糖開關具備可與ON/OFF比500相媲美的全球最高性能。據悉,在相關領域中,三位數的ON/OFF比尚無先例。
此外,研究團隊利用開發的兩種核糖開關,構建了調控γ-溶血素功能表達的邏輯電路(與閘),並證實該電路可正常運行。
據悉,未來研究團隊將進一步提升ON/OFF比,並驗證RNA-RBP配對能否在大腸桿菌等不同生物平台中發揮作用。
福永副教授表示:「此次,我們成功解析了通過人工進化(PD-SELEX)獲得的超高親和性RNA-蛋白質(RNP)配對結構,並開發出利用RNP元件的無細胞系統高性能翻譯開關。今後,我們還將致力於開發可在細胞內發揮作用的RNA器件。」
原文:《科學新聞》
翻譯:JST客觀日本編輯部
【論文資訊】
期刊:Nucleic Acids Research
論文:Structural insights into lab-coevolved RNA-RBP pairs and applications of synthetic riboswitches in cell-free system
DOI:10.1093/nar/gkaf212