東京大學研究生院農學生命科學研究科岩田洋佳教授等通過整合以及聚類分析植物根圈土壤或動物腸道等寄主生物周邊的微生物組(Microbiome)與代謝物組(Metabolome)的數據,開發出了能夠明確微生物與代謝物關係的「整合概率變分變量選擇」(I-SVVS)新方法。該方法有望深化理解多種寄主生物中微生物組與代謝物組的相輔作用,推動農業、醫療及環境科學發展。相關研究成果已發表在《Briefings in Bioinformatics》上。
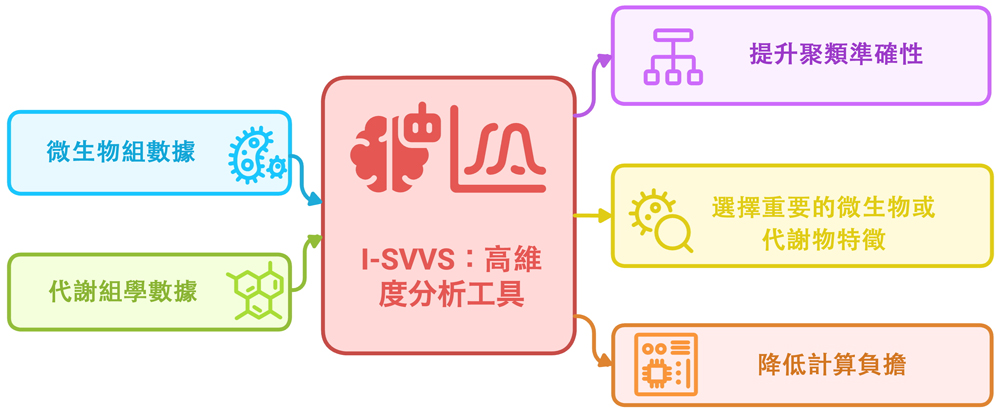
圖1 用於多組學微生物組研究的I-SVVS解析法(供圖:東京大學)
多組學微生物組數據是指用於理解微生物群集功能及相輔作用,整合宏基因組(表徵微生物種類的遺傳資訊)以及代謝組等各類組學技術的統合數據。這類數據具有資訊量大,但數據點密度低、多個變量強相關(多重共線性)的特點,需要高級計算方法才能準確解析。
研究團隊開發的I-SVVS法是首個專用於高維微生物組數據分析的方法。I-SVVS法採用基於概率變分推斷的變量選擇框架,對微生物數據應用無限狄利克雷多項分布混合模型,對代謝物數據應用無限高斯混合模型,從而提升計算效率與解析精度。此外,該方法能從數據內的海量特徵中識別出包含關鍵資訊的最小特徵集,實現高精度分類(聚類)並發現微生物與代謝物的關聯性。
使用大豆、小鼠和人類三組數據集進行的實驗結果表明,I-SVVS在所有案例中均展現出比既往解析方法(iClusterPlus和Clusternomics)更高的精度與更快的處理速度。特別是在大豆數據集(377個樣本、16,943種微生物、265種代謝物)中表現出更快速精準的解析能力。成功鑑定出與大豆生長相關的關鍵微生物群,並闡明瞭不同環境條件下微生物與代謝物的關聯性。
上述結果表明I-SVVS有望大幅推動多組學微生物組研究。此前,難以明確微生物功能與寄主健康、生長的關係,解析複雜數據需耗費大量時間和精力。而使用I-SVVS法能更準確理解微生物如何與寄主生物相輔作用,如何促進生長或引發疾病。
例如,該方法可詳細解析土壤微生物如何促進植物生長,或腸道菌群如何影響人類健康。這在農業領域將有助於提高作物產量、推動可持續農業發展,在醫療領域可促進基於腸道菌群的疾病預防與治療方法的開發。在環境領域也能深化理解微生物對生態系統平衡的作用,為自然環境保護和應對氣候變化做出貢獻。
未來通過利用I-SVVS法,有望進一步闡明微生物與寄主之間的關係,並在農業、醫療、環境科學等廣泛領域實現應用。
原文:《科學新聞》
翻譯:JST客觀日本編輯部
【論文資訊】
期刊:Briefings in Bioinformatics
論文:I-SVVS: Integrative stochastic variational variable selection to explore joint patterns of multi-omics microbiome data
DOI:10.1093/bib/bbaf132