日本九州工業大學和理化學研究所通過共同研究開發出一種新型資訊科技,能高精度預測各種人體細胞中藥物反應的基因表現模式(圖1)。
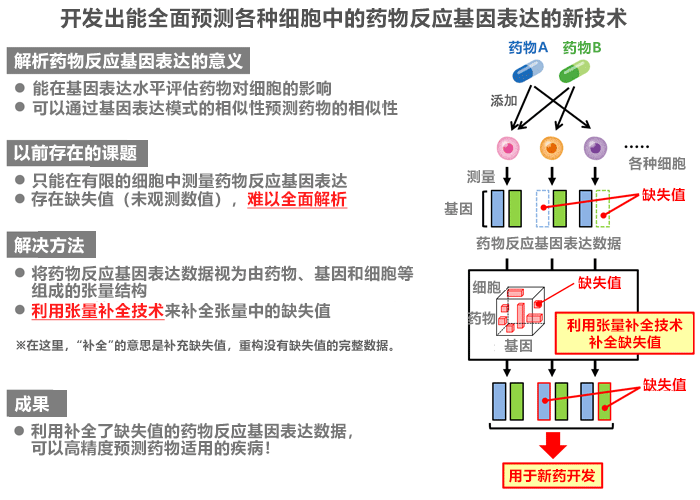
圖1:發布概要
本次研究將藥物反應基因表現數據視為由藥物、基因、細胞和時間序列構成的張量結構,開發了機器學習方法,利用新的張量分解法Tensor Train Weighted Optimization演算法,來預測各種細胞中尚未觀測到的藥物反應。研究小組利用新開發的方法預測了藥物的療效,結果表明,這種方法有望大大提高療效預測的性能。
首先,為了評估由藥物、細胞和基因構成的3階張量結構——藥物反應基因表現數據中的未觀測值預測性能,研究小組將觀測值人工改為缺失值,調查了能否準確再現觀測值。結果發現,新方法與以往的方法相比,最大能以約1.5倍的精度補全缺失值(圖2)。另外,由於藥物反應基因表現模式會隨著時間變化,因此研究小組構建了由藥物、細胞、基因和時間序列組成的4階張量,實施了相同的性能評估。結果顯示,新方法對高階張量結構的數據尤其有效(精度與以往的方法相比最大提高約2倍)。
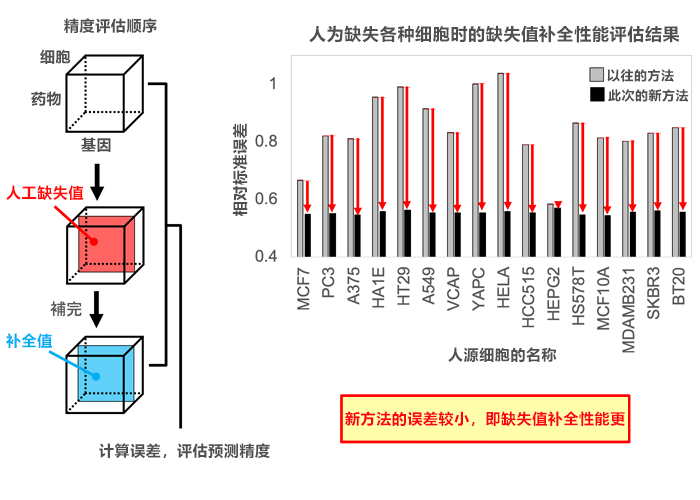
圖2:新方法與以往的方法相比,補全張量缺失值的性能更高
接下來,研究小組將補全了缺失值的藥物反應基因表現數據應用於藥物的療效預測。結果顯示,缺失值越多的細胞,越能大幅提高療效預測的性能,性能最大提高約20%(圖3)。另外,研究小組把新提出的方法應用於將1483種藥物添加到16種人體細胞中獲得的藥物反應基因表現數據,篩選出了對很多疾病均有望發揮療效的藥物,比如驅蟲藥氯硝柳胺能治療成人T細胞白血病等。此外,還通過近年來的文獻和臨床報告,確認了新預測的藥物療效的有效性。
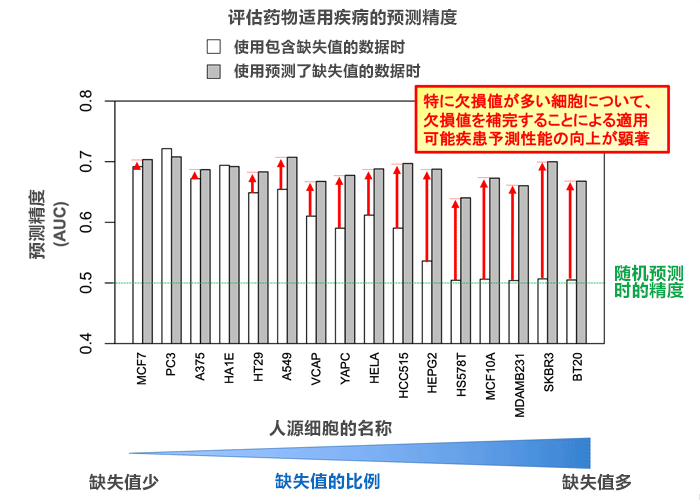
圖3:通過新提出的方法補全了缺失值的基因表現數據對新藥開發也有用
利用這種可預測各種人體細胞中的藥物反應的新方法,有望促進新藥開發,包括查明藥物的作用機制、預測藥效以及探索工具化合物等。今後計劃進一步改善演算法,提高預測可靠性和計算效率。同時,不僅是已經獲批的藥物,還將把解析對象擴大到合成化合物和天然化合物等所有化合物中,以探索針對各種疾病的藥物候學選化合物。
(日文新聞發布全文)
文:JST客觀日本編輯部翻譯整理