熊本大學人類逆轉錄病毒學共同研究中心的佐藤賢文教授與佐賀大學勝屋弘雄助教、鹿兒島大學宇都宮與客座教授、聖瑪麗醫科大學山野嘉久教授等共同確立了全新的解析方法,能夠全方位高精度獲取被HTLV-1(人類T細胞白血病病毒)感染細胞中插入的病毒DNA資訊。
日本目前約有HTLV-1感染者80萬人左右,已知部分感染者會轉化成癌症或慢性發炎等其他疾病。HTLV-1病毒感染細胞後會將自己的部分DNA插入整合到寄主細胞DNA中。整合到寄主細胞中的病毒,是這些相關疾病的根本性病原。所以,獲取已整合進的病毒DNA資訊對相關疾病的診斷及發病機理研究都具有極為重要的意義。
一直以來,為了準確掌握被HTLV-1感染患者的病毒感染病理,需要結合多種解析方法來獲取病毒DNA序列全長(判定是否有缺陷型)、病毒DNA的結構、病毒DNA插入位點、被感染細胞的增殖情況等資訊。此次研究組建立的全新技術,整合了病毒DNA獨特性探針和下一代測序技術,從而實現一次性對病毒資訊進行全面高精度的解析(圖1)。
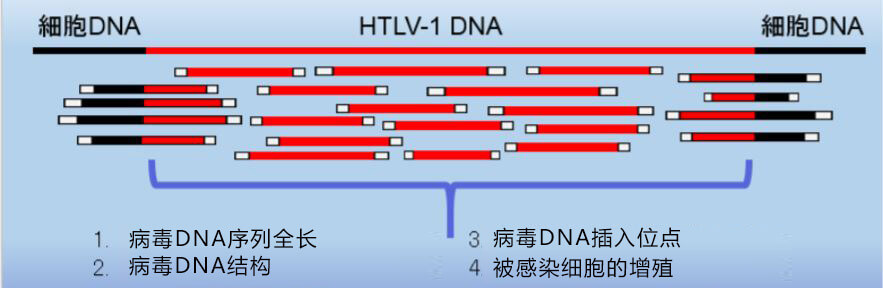
圖1 全面高精度解析HTLV-1 DNA的新技術
研究團隊進一步採用該新技術,解析了100名日本被感染患者的詳細病毒相關資訊。除了撰寫成論文公開發表,上述病毒資訊資源也一併上傳到公共數據庫以供查閱,希望對後續HTLV-1的研究及相關疾病的診斷有所貢獻。
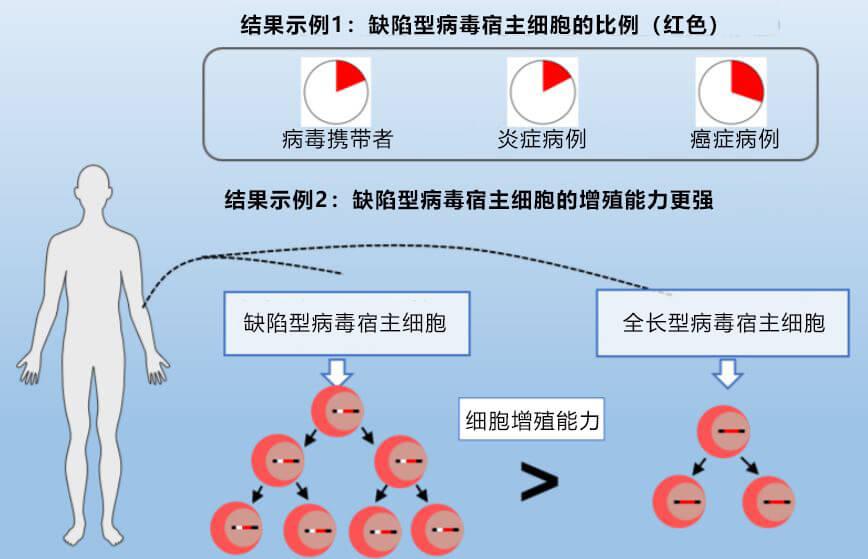
圖2 通過新技術獲取的病毒相關資訊示例
採用DNA探針和下一代測序技術結合的新方法,不僅適用於HTLV-1的傳染病,而且可以依此類推應用於其他癌症相關的病毒傳染病領域。本研究成果已於2019年10月15日發表在《Cell Reports》上[文獻1]。
參考文獻:
1. Katsuya H etc. The nature of HTLV-1 provirus in naturally infected individuals analyzed by viral DNA-capture-seq approach.
Cell Reports. 2019 Oct 15;29(3):724-735.e4.
DOI: 10.1016/j.celrep.2019.09.016.
相關鏈結:
日文新聞發布全文
文:JST客觀日本編輯部翻譯整理