日本東京工業大學資訊理工學院資訊工學系的關嶋政和副教授,該校物質與資訊卓越教育院的安尾信明特任講師和筑波大學醫學醫療系生命醫科學領域的吉野龍之介助教組成研究團隊,利用超級計算機TSUBAME3.0進行分子動力學模擬,成功建立了抑制參與新冠病毒(SARS-CoV-2)克隆的「主蛋白酶」功能的治療藥物候學選化合物應該滿足的藥效團模型。另外,還利用靶向主蛋白酶的候選藥物α-酮酰胺抑制劑(參考文獻)確認該藥效團是正確的。
尋找新冠病毒感染症(COVID-19)治療藥物時主要利用藥物重定位法,即以現有藥物或者通過了部分臨床試驗的藥物為基礎進行探索,這樣可確保安全性並縮短開發時間。此次研究確認的藥效團不僅可用於現有藥物的探索,還可以用來探索新的藥物候學選化合物,就這一點而言,可以說是一項重要的成果。
今後計劃以該藥效團為基礎,通過結合了模擬及統計機器學習等人工智慧(AI)和生物化學實驗的智慧新藥開發,探索能成為新冠病毒感染症(COVID-19)的候選治療藥物的具體化合物。
目前發現的事實是,新冠病毒最早於2019年12月在中國武漢出現,隨後引起大流行。截至2020年8月下旬,新冠病毒感染症的感染人數已經超過2300萬人,死亡人數超過80萬,目前感染仍在持續擴大之中,全球都在尋找治療藥物。以前有報告顯示,肽樣抗HIV-1藥物能有效抑制參與SARS病毒(SARS-CoV)克隆的主蛋白酶。由於SARS-CoV與SARS-CoV-2之間有密切的系統關聯,主蛋白酶在結構和功能上有很多共同特徵(圖1)。因此,肽樣抗HIV-1藥物也被認為是靶向SARS-CoV-2的主蛋白酶的候選藥物,不過一直不清楚SARS-CoV-2的主蛋白酶在原子水平上的作用機制。
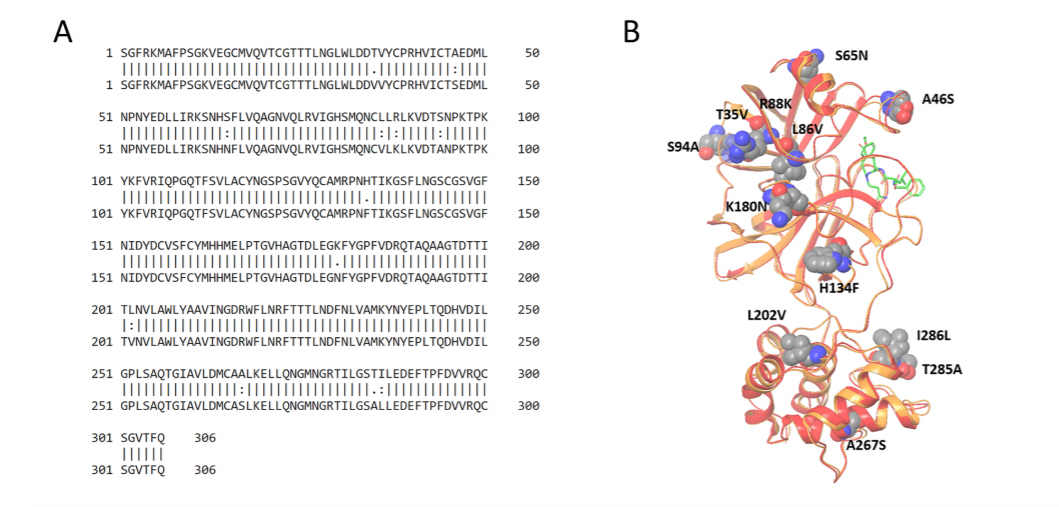
圖1:SARS病毒(SARS-CoV)與新冠病毒(SARS-CoV-2)的主蛋白酶胺基酸序列和蛋白質空間結構的比較。胺基酸序列有96%相同。
此次研究團隊利用超級計算機TSUBAME3.0在1微秒的時間尺度上實施分子動力學(MD)模擬,明確了SARS-CoV-2的主蛋白酶與3種藥物候學選化合物之間的重要相輔作用,並成功建立了藥效團模型。在所有的MD模擬中,SARS-CoV-2主蛋白酶的第41位His(組胺酸)、第153位Gly(甘胺酸)和第166位Glu(麩醯胺酸)都與化合物的同一個官能基相輔作用。這個結果表明,相輔作用是作用於SARS-CoV-2主蛋白酶的藥物的重要靶標。
為驗證建模的藥效團的正確性,研究團隊調查了已知能以IC50=0.67±0.18μM抑制SARS-CoV-2主蛋白酶功能的α-酮酰胺抑制劑是否符合該藥效團。結果確認,α-酮酰胺的一個羥基和兩個羰基與藥效團模型一致(圖2)。
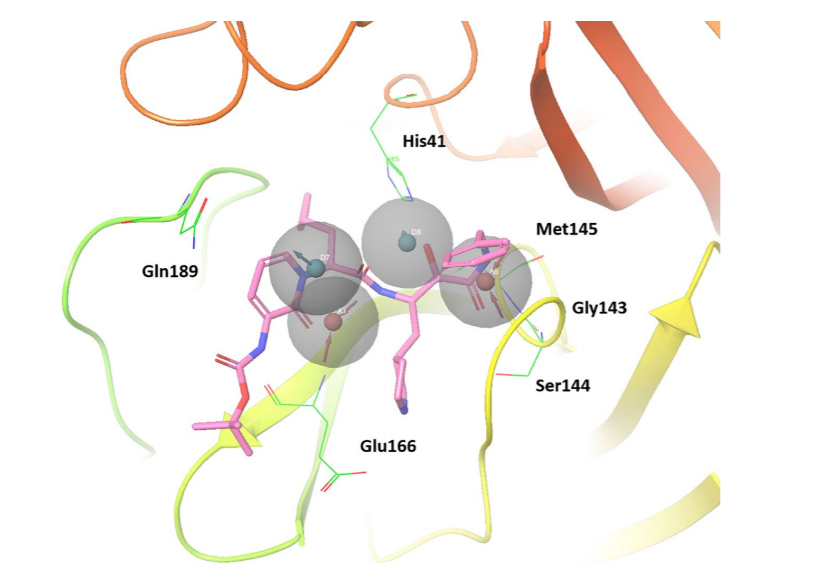
圖2:圍繞SARS-CoV-2主蛋白酶和候選藥物α-酮酰胺抑制劑,驗證是否符合本次研究建模的藥效團
SARS病毒(SARS-CoV)有很多與主蛋白酶的第145位Cys(半胱胺酸)共價結合的不可逆性抑制劑,這些抑制劑可能具有比競爭性抑制劑更高的結合親和性。不過,對於COVID-19等高度緊急的疾病來說,藥物重定位比較有效,為了與Cys形成共價鍵,此次研究提出的藥效團可以評估不含官能基的化合物。
【參考文獻】
Zhang et al., Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors,Science 24 Apr 2020:Vol. 368, Issue 6489, pp. 409-412,DOI: 10.1126/science.abb3405
論文資訊
論文題目:Identification of key interactions between SARS-CoV-2 mainprotease and inhibitor drug candidates
發表期刊:Scientific Reports
DOI:10.1038/s41598-020-69337-9
文:JST客觀日本編輯部