本文根據理化學研究所成果發布編譯而成
由日本理化學研究所(簡稱「理研」)計算科學研究中心粒子系生物物理可用能研究團隊的杉田有治擔任組長的研究團隊,利用超級計算機「富嶽」和「Oakforest-PACS」對新冠病毒(SARS-CoV-2)表面存在的「刺突蛋白」進行的模擬計算發現,修飾刺突蛋白表面的糖鏈在病毒侵入人體細胞時發生的刺突蛋白結構變化中發揮著重要作用。
2020年12月在英國發現的突變病毒株中存在17處突變,傳染性比以前更強。全球各國都在對刺突蛋白進行分子動力學模擬,利用「富嶽」模擬計算獲得的蛋白質詳細結構資訊可以為藥品開發提供線索。利用「富嶽」和「Oakforest-PACS」能進行高速分析,對於突然出現的突變病毒株等問題,也可以利用超級計算機迅速應對,由此有望為預防和治療新冠病毒感染症(COVID-19)的藥物的分子設計做出貢獻。
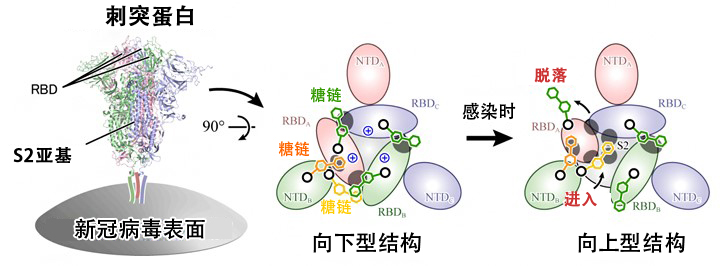
新冠病毒表面存在的刺突蛋白結構變化機制
在新冠病毒進入人體細胞的初期階段,病毒表面存在的蛋白質(刺突蛋白)會與人體細胞表面的血管收縮素轉化酶II(ACE2接受者)結合併吸附到細胞上面,隨著病毒侵入發生感染(圖1A)。刺突蛋白由3條多肽鏈組成,每條多肽鏈由N端結構域(NTD)、接受者結合結構域(RBD)和S2結構域構成(圖1B左)。生物化學實驗還顯示,刺突蛋白表面的很多氨基酸都是由糖鏈修飾的。不僅是刺突蛋白,很多蛋白質的表面都通過翻譯後修飾增加了糖鏈,研究認為,蛋白質相互識別時就利用這些糖鏈。另外,就病毒而言,還有觀點認為,刺突蛋白為躲避抗體的攻擊而被糖鏈包裹住了。
研究團隊利用X射線晶體結構分析法和冷凍電子顯微鏡進行單顆粒分析發現,新冠病毒的刺突蛋白具有「向下型」(Down型)和「向上型」(Up型)兩種結構(圖1B)。在向上型結構中,RBD會從S2次單元移開(圖1B紅色部分)。另外還發現,刺突蛋白不與ACE2接受者結合時,會在向下型和向上型之間來回變化(平衡狀態),與ACE2接受者結合後,向上型會變得更穩定。
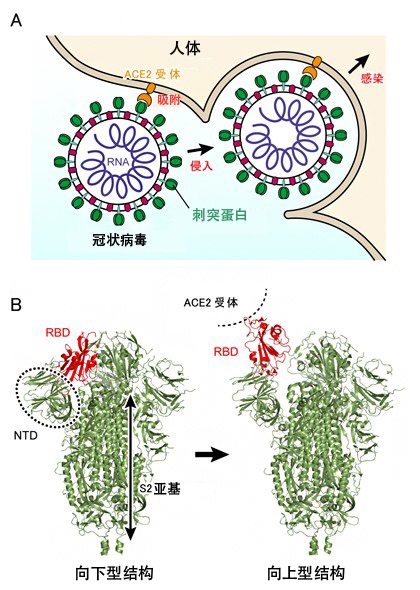
圖1:新冠病毒SARS-CoV-2的感染機制
研究團隊為調查刺突蛋白的詳細分子結構,利用超級計算機「富嶽」和「Oakforest-PACS」,對向下型結構和向上型結構進行了分子動力學模擬(圖2A)。分子動力學模擬是通過在計算機中構築虛擬分子系統,並求解每個原子的牛頓運動方程式F=ma,來從理論上預測穩定的分子結構,以及視覺化分子運動的方法(圖2B)。分子系統非常大時,運算量也很龐大,因此需要利用超級計算機進行高速計算。
研究團隊2015年自主開發出了分子動力學計算軟體GENESIS。GENESIS被選為「富嶽」統合設計開發的目標應用之一,Jaewoon Jung專任工程師和小林千草工程師等人主導針對「富嶽」進行了最適化,應用執行性能達到「京」的100倍以上。此次利用GENESIS,對刺突蛋白進行了1微秒(100萬分之1秒)的分子動力學計算,全面分析了RBD結構變化之前和之後的氨基酸-氨基酸及糖鏈-氨基酸之間的相輔作用。
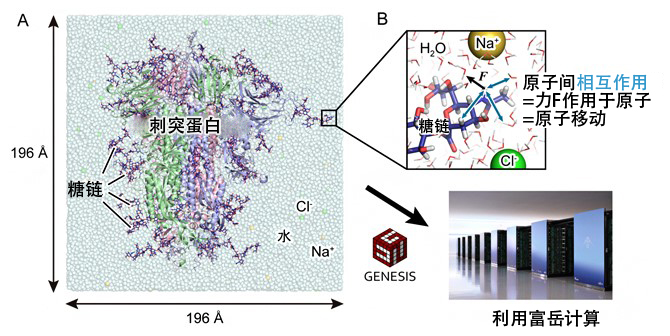
圖2:利用GENESIS和「富嶽」模擬SARS-CoV-2的刺突蛋白
由此發現,修飾刺突蛋白第165位、第234位和第343位三個天冬醯胺酸N165、N234和N343的各糖鏈在RBD的結構穩定中發揮著重要作用。在向下型結構中,N343和N165的糖鏈分別連接RBD-RBD(圖3A上)和RBD-NTD,從上方覆蓋住了RBD(圖3B上)。變為向上型時,N343的糖鏈脫落(圖3A下),然後進入N234的糖鏈變為向上型時產生的RBD-S2結構域之間的空腔(圖3C),並與S2結構域和RBD形成強氫鍵,以從下面強化移動到上部的RBD(圖3B下)。
另外,對向下型結構實施靜電位分析發現,三個RBD的邊界面通過第378位離胺酸(K378,圖3A中央藍色)和第407位精胺酸(R407)等大範圍帶正電。
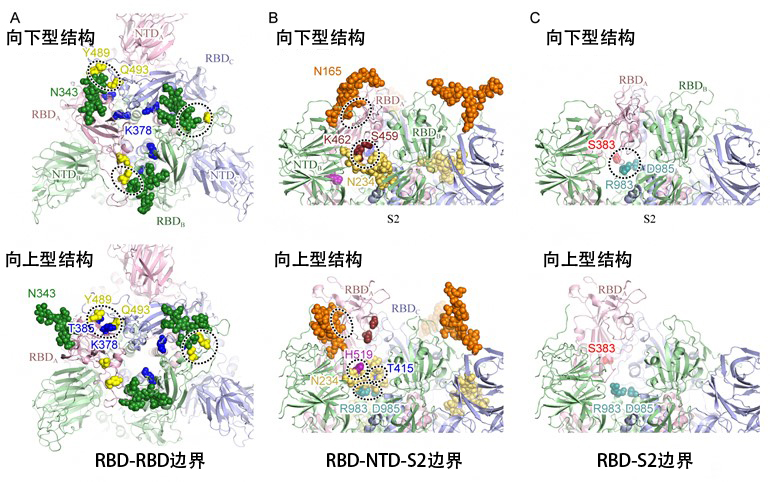
圖3:刺突蛋白中的氨基酸-氨基酸及氨基酸-糖鏈相輔作用
這些結果表明,結構朝向上同質多相變體化是以RBD間的靜電斥力為驅動力發生的,另外,穩定向下型結構的糖鏈脫落,RBD移動後,其他糖鏈會進入(圖4)。研究團隊不僅通過此次研究明確了新冠病毒感染的詳細分子機制,還成功發現了糖鏈穩定蛋白質動態結構的新生物學知識。
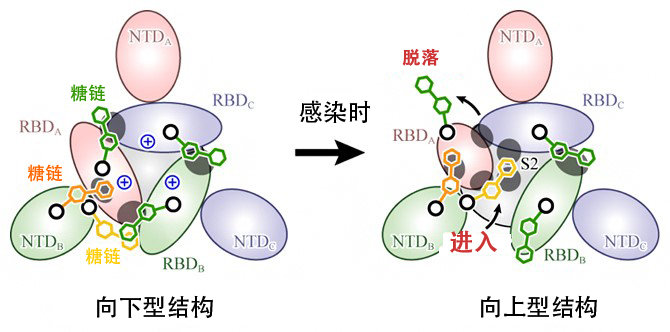
圖4:刺突蛋白的結構變化機制
刺突蛋白的俯視圖。由向下同質多相變體為向上型的結構變化以RBD間的正電荷(中央藍色+標記)形成的靜電斥力為驅動力發生,穩定向下型結構的糖鏈(綠色)脫落,RBD移動後,其他糖鏈(黃色)進入,穩定向上型結構。
研究團隊此次的發現著眼於在刺突蛋白的結構變化中發揮增強作用的糖鏈,有望創造新的治療戰略,例如「防止向下型的RBD-RBD間的糖鏈脫落」,或者「抑制變為向上型時進入的糖鏈」等。
論文資訊
題目:Elucidation of interactions regulating conformational stability and dynamics of SARS-CoV-2 S-protein
期刊:Biophysical Journal
DOI:10.1016/j.bpj.2021.01.012
日語發布資料
編譯:JST客觀日本編輯部