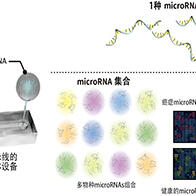
日本理化學研究所(簡稱理研)BDR(Center for Biosystems Dynamics Research)中心的金堅石博士和城口克之教授等與京都大學合作,成功開發出了名為BarBIQ(Barcoding Bacteria for Identification and Quantification)的高通量菌群分析新技術。該技術基於16S rRNA基因測序和單細胞標籤等技術,實現了對菌群中單細胞(細菌個體)的精確鑑定和細胞數的定量,對單細胞中16S rRNA序列的鑑定可以同時達到單鹼基正確率和單鹼基解析度,是目前唯一可以同時做到既見「森林」(菌群整體)又見樹木(細菌個體)的菌群分析方法,開創了菌群研究領域的先河(圖1)。該技術未來可用於腸內菌群相關疾病的理解和診斷,還可以高精度測量皮膚、口腔、植物、土壤、海洋以及大氣中存在的菌群,具有廣泛的科學用途。相關内容已經發布在《Nature Communications》線上版上(2月22日)。
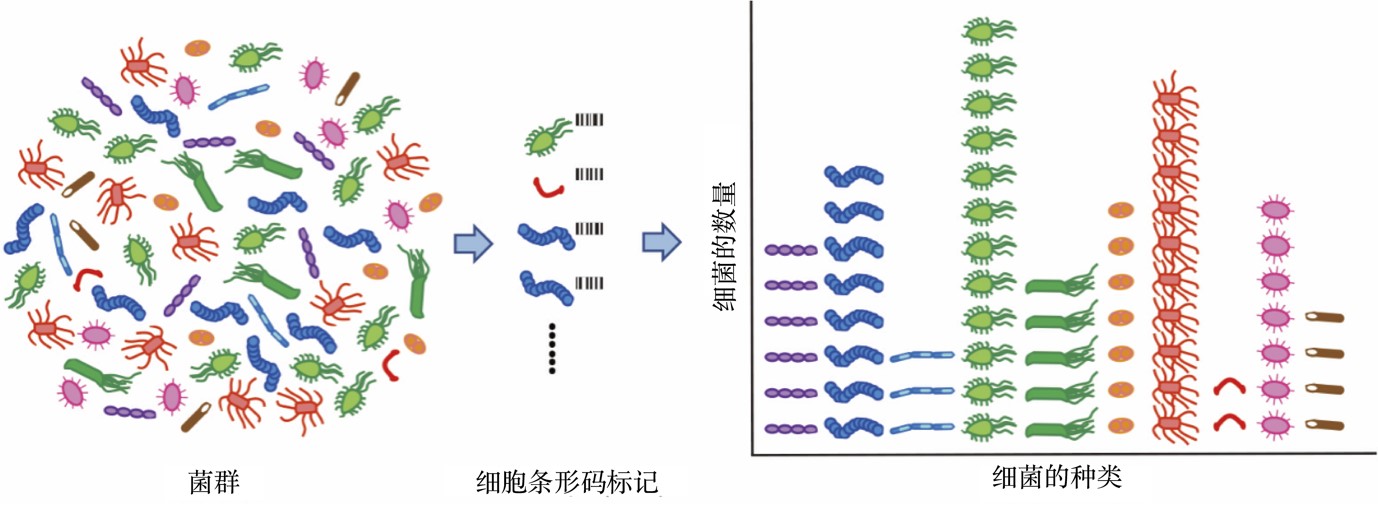
圖1 單細胞菌群分析方法示意圖
人類的腸道居住著約500~1000種細菌,近100萬億個細菌,這種細菌組成的群體被稱為「菌群」。腸內菌群的平衡與腸内免疫以及健康密切相關,所以準確測量各種細菌的分佈情況是極為重要的。此次,該研究團隊將細菌逐一封入液滴,並對每個細菌的16S rRNA基因標記上不同的被稱為條形碼的DNA序列,從而實現了一次性對10萬個以上的細菌的種類的鑑定和各種類細菌的定量。利用這種方法,他們精確測定了小鼠經維他命A缺乏食物餵養後其盲腸內菌群的微小變化。
BarBIQ方法是在城口克之等人先後於2012年和2017年發表的DNA分子條形碼標記法基礎上開發成功的。該方法將菌群中的細菌單細胞一個個封裝入液滴,並在DNA擴增中對每個細菌細胞的16S rRNA基因標記上唯一的DNA系列(這種策略也被稱為細胞條形碼標記)。具體操作如下。
① 菌群中的細菌單細胞和一個具有唯一序列的單分子DNA(細胞條形碼)被共封裝入液滴 (圖2)。
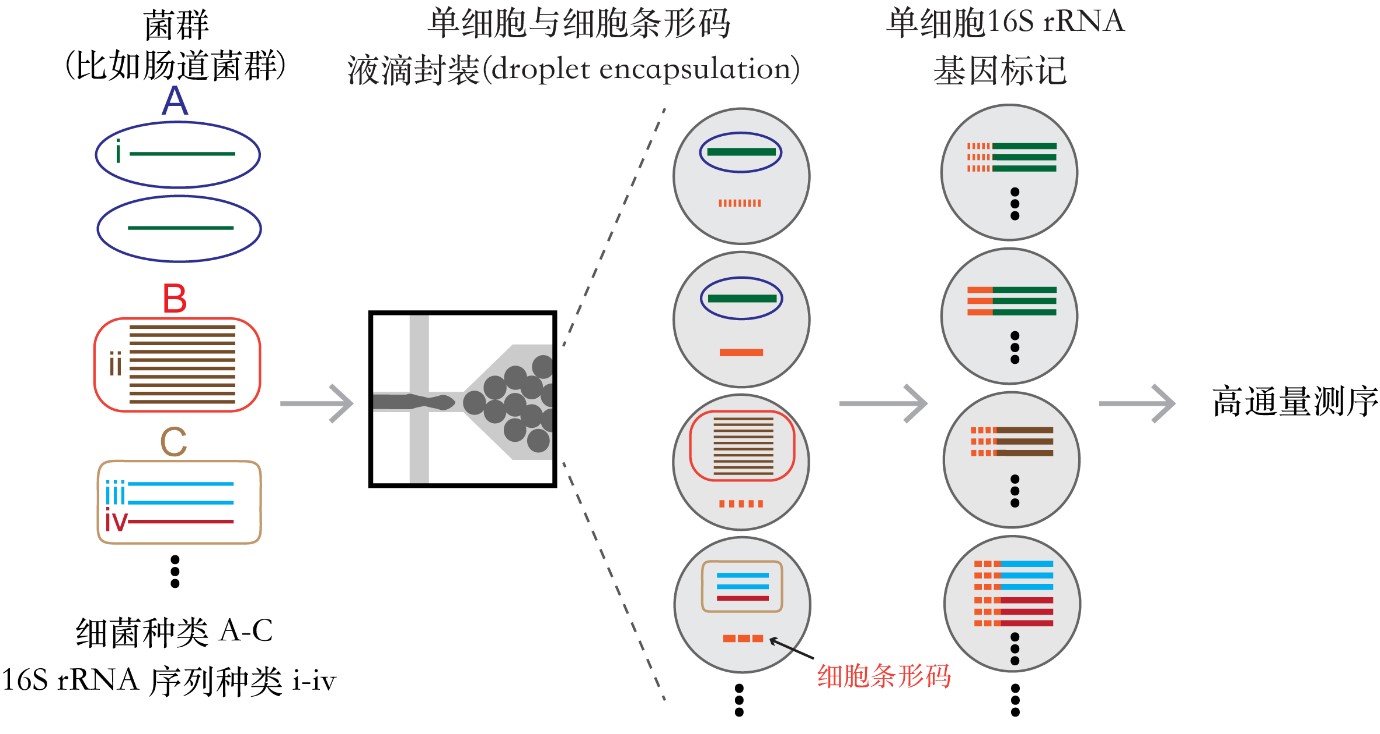
圖2 BarBIQ法示意圖
② 細菌中的16S rRNA基因被擴增並和同一液滴中的細胞條形碼相連接。因此不管來自同一個細菌的16S rRNA基因有多少個或者多少種,都將被標記上相同的細胞條形碼(圖2)。這是實現單細胞分析的關鍵步驟。
③ 液滴中所有被擴增的DNA被純化和二代測序分析後,16S rRNA基因序列和細胞條形碼會被成對讀出。之後,16S rRNA基因序列被用來鑑定細菌的種類,而細胞條形碼則被用來確定這些16S rRNA基因序列來自哪個細胞。
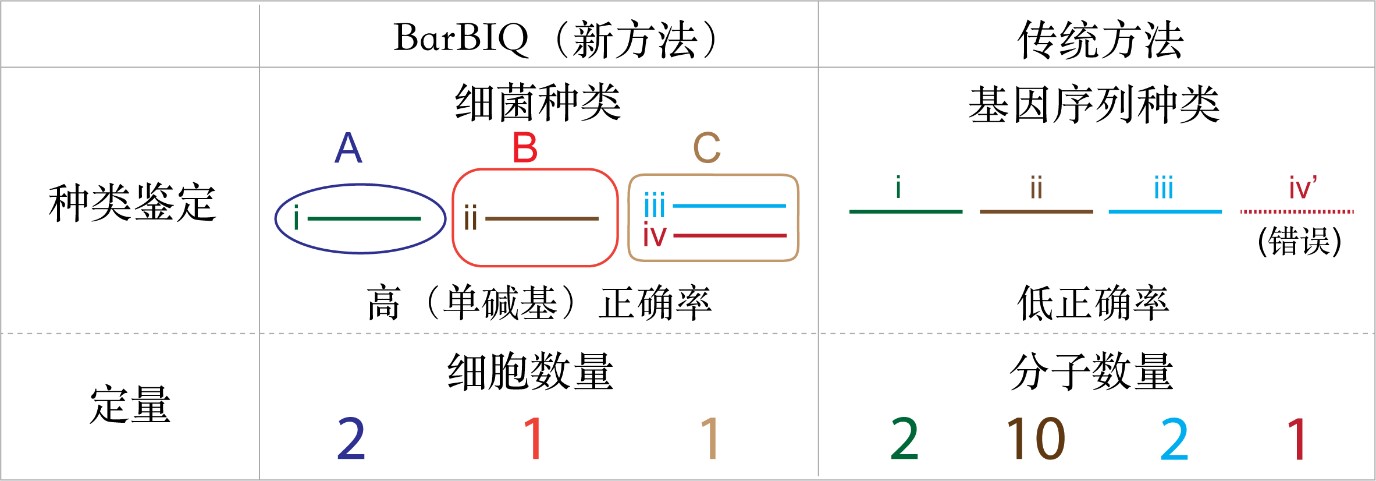
圖3 BarBIQ法與傳統方法的比較
16S rRNA基因在不同種類細菌中具有不同的序列,因此被廣泛用來鑑定細菌的種類。BarBIQ方法在DNA擴增時,每個細菌細胞都被隔離在一個液滴中,因此不同細菌的16S rRNA基因不會被嵌合擴增成不準確的DNA產物,而大量擴增出來的DNA嵌合體正是造成傳統方法鑑定16S rRNA序列不準確的主要原因之一。
另外,BarBIQ方法中每個細菌被不同的細胞條形碼標記,所以哪怕只有一個鹼基不同的16S rRNA序列,也能被鑑別出是來自不同細菌的還是來自同一細菌的相似序列。因此,BarBIQ方法相比於傳統方法,首次實現了基於細胞的16S rRNA序列鑑定,同時還具有單鹼基準確率和單鹼基解析度(圖3)。
此外,因為每個細菌個體都是被不同細胞條形碼標記的,即使是相同的細菌也能通過被標記的條形碼區分開,因此BarBIQ方法可一次性鑑定超過10萬個細菌的種類還能對每一種鑑定細菌進行細胞計數。而傳統方法無法追蹤每個細胞,因此只能實現對被擴增16S rRNA分子的鑑定和計數(圖3)。

圖4 封入細菌的液滴
左為剛做好的液滴明場顯微鏡圖像;右為液滴中細菌DNA經螢光染色後的螢光圖像。箭頭所指處為觀察到的細菌
論文:
Jianshi Jin, Reiko Yamamoto, Tadashi Takeuchi, Guangwei Cui, Eiji Miyauchi, Nozomi Hojo, Koichi Ikuta, Hiroshi Ohno, Katsuyuki Shiroguchi, High-throughput identification and quantification of single bacterial cells in the microbiota, Nature Communications (2022) doi.org/10.1038/s41467-022-28426-1.
文 曹暉 JST客觀日本