日本名古屋大學研究生院工學研究科的清中茂樹教授、京都大學研究生院工學研究科的浜地格教授及慶應義塾大學醫學部的柚崎通介教授等人組成的研究團隊宣佈,開發出了可以選擇性激活麩胺酸受體的「配位化學遺傳學方法」,通過向支持運動功能和運動學習的小腦神經網路中發揮作用的代謝型麩胺酸受體1型(mGlu1)導入突變,可以在保留對原始配體麩胺酸的反應功能的同時,對人工配體做出反應後被激活。該成果有助於明確各種神經細胞的功能,並開發與神經活動異常有關的疾病的治療方法。相關内容已經發布在國際科學期刊《Nature Communications》的6月16日號上。
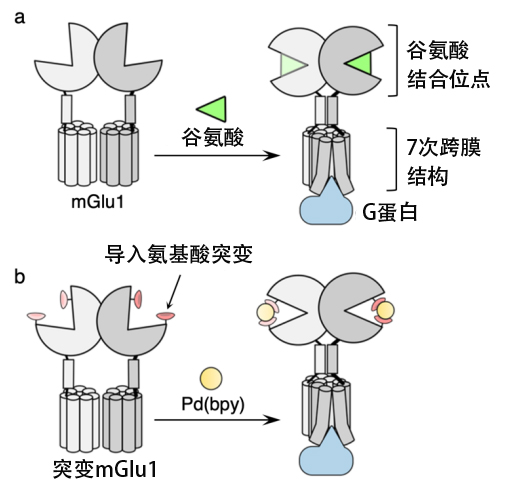
圖1:通過配位化學遺傳學方法激活mGlu1。(a)mGlu1的結構以及通過麩胺酸結合進行激活的模式圖,(b)通過配位化學遺傳學方法激活mGlu1的模式圖(供圖:名古屋大學)
腦内的大量神經細胞錯綜複雜地連接在一起,形成神經網路,從而實現高級的腦功能。研究人員認為,要了解腦功能,就需要明確各種神經細胞的功能,儘管腦内不同類型的神經細胞功能各不相同,但都是通過多個共同的接受者來傳遞資訊的。因此,即使使用針對各種接受者的活化劑等,也很難明確每種接受者的功能。
對此,研究人員近年來開發了選擇性激活靶細胞類型的光遺傳學和化學遺傳學等方法,但這些方法都是通過強製表達人工蛋白質來控制細胞活性,因此無法分析腦内的實際神經傳遞。光遺傳學可以通過基因導入光響應蛋白,利用光來操控細胞活性,但需要向腦内物理可用能植入光纖,侵襲性較高,而且不能應用於腦深處光無法到達的細胞。此外,化學遺傳學可通過在細胞中表達用不作用於體內蛋白質的人工配體選擇性激活的人工接受者來操控細胞活性,但接受者的位置與原來不同。
為此,研究團隊此次著眼於負責運動功能和運動學習、主要在小腦的多種神經細胞中豐富表達的mGlu1。為明確每種細胞的功能,研究了用人工配體選擇性激活mGlu1的方法,即「配位化學遺傳學方法」。
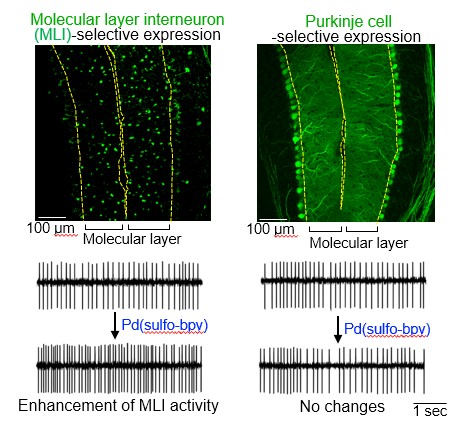
圖2:通過配位化學遺傳學方法進行細胞選擇性的mGlu1激活。(上圖)利用腺相關病毒確認MLI及普金斯細胞選擇性基因表現(通過綠色螢光蛋白確認),(下圖)MLI的自發神經活動。利用Pd(sulfo-bpy)激活MLI的mGlu1時,確認神經活動增加。(供圖:名古屋大學)
研究發現,mGlu1在細胞外有麩胺酸結合位置,麩胺酸(天然配體)結合通過該位點的結構變化將資訊傳遞到細胞内。
因此,為通過人工配體結合引起這種結構變化,研究團隊在麩胺酸結合位置導入突變,探索了與之配對分組的人工配體。具體來說,在麩胺酸結合位置的入口處導入組胺酸(金屬配位氨基酸)突變,發現了與之配對分組的人工配體鈀聯吡啶錯合物(Pd(bpy))。向突變mGlu1施用人工配體確認,可以在維持常規麩胺酸反應的同時,對人工配體也做出了反應。
研究團隊還進一步評估了人工配體的細胞毒性,設計了不影響細胞增殖和神經突起延伸的人工配體Pd(sulfo-bpy)。
研究團隊通過基因體編輯(CRISPR/Cas9)將這種突變導入小鼠體內確認,小鼠的運動和其他功能都顯示正常。
向相同小鼠的小腦剖切中添加Pd(sulfo-bpy)後,觀察到了通常由mGlu1的激活引起的小腦長期抑制(突觸電流反應持續減弱),而為野生小鼠添加該人工配體則未觀察到這種現象。
在小腦中,mGlu1不僅在普金斯細胞中表達,還在調節普金斯細胞功能的顆顆粒球及分子層中間神經(MLI)中也有表達。關於MLI此前有報告顯示,向小腦剖切施用mGlu1活化劑(DHPG)會增強神經活動,但一直不清楚這種增強對哪種神經細胞的激活比較重要。
對此,研究團隊此次利用新開發的方法,通過腺相關病毒和細胞類型選擇性基因表現啟動子在小鼠腦内的普金斯細胞和MLI中分別表達了突變mGlu1。然後向這些小鼠的小腦剖切施用Pd(sulfo-bpy)。
結果顯示,神經活動在MLI中增強,但在普金斯細胞中沒有增強,由此首次確認,MLI的活動增強依賴於MLI中表達的mGlu1的激活。
該方法被認為不僅適用於mGlu1,也適用於其他麩胺酸受體,還有望加速闡明與麩胺酸受體有關的記憶和學習等高級腦功能,並作為由這些接受者異常引起的神經疾病的治療方法使用。
清中教授表示:「mGlu1被認為對運動學習非常重要。今後將為在普金斯細胞中表達突變mGlu1的小鼠添加人工配體,觀察小鼠的行為會出現什麼樣的表型,以及運動學習能力是否會得到改善。瞭解哪些大腦迴路在控制人類的行為是新一代神經科學的研究範疇。」
原文:《科學新聞》
翻譯編輯:JST客觀日本編輯部
【論文資訊】
期刊:Nature Communications
論文:Coordination chemogenetics for activation of GPCR-type glutamate receptors in brain tissue
DOI:10.1038/s41467-022-30828-0