日本自然科學研究機構生命創成探究中心的加藤晃一教授與名古屋市立大學藥學研究科的矢木宏和副教授等人組成的研究團隊著眼於由特定糖鏈修飾的蛋白質,成功發現了其分子結構中應稱之為糖鏈修飾控制碼的胺基酸序列。另外還證明了通過編入該分子碼,可以對生物藥品中的蛋白質實施特定的糖鏈修飾。
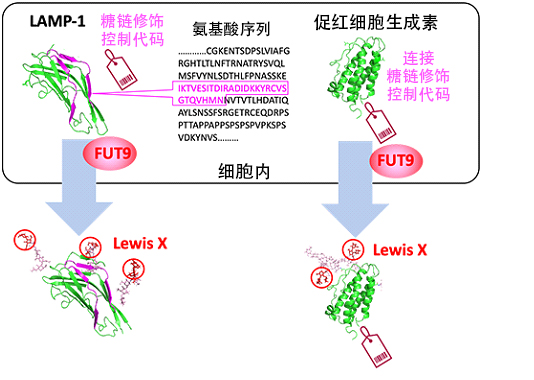
圖1:在LAMP-1的n結構域中發現了負責Lewis X修飾的特定29個殘基的胺基酸序列,另外還發現了編入了這種糖鏈修飾碼的促紅血球生成素能通過FUT9促進對Lewis X的修飾(供圖:自然科學研究機構生命創成探究中心、名古屋市立大學)
蛋白質是以基因為設計圖形成的,而糖鏈沒有明確的設計圖。研究團隊認為,蛋白質的胺基酸序列(基因資訊)中可能隱藏著糖鏈的部分設計圖。此前的研究發現,在小鼠神經幹細胞中,具有Lewis X結構的糖鏈會獨特性修飾名為LAMP-1的蛋白質。這種Lewis X結構是在岩藻糖基轉移酶9(FUT9)的作用下形成的。
研究團隊此次發現,Lewis X對LAMP-1的獨特性修飾不僅是神經幹細胞,還發生在幾種培養的動物細胞中。LAMP-1由形狀非常相似的兩個結構域(N結構域和C結構域)組成,但使其分別在細胞中表達時發現,僅N結構域會被Lewis X修飾。
研究團隊利用兩個結構域相似的特點,製備了一系列兩個結構域分別被部分更換的蛋白質,比較這些蛋白質的糖鏈修飾發現,N結構域中存在由29個胺基酸殘基構成的序列,由此在其周邊部位通過FUT9引發了Lewis X修飾。另外還發現,隻需將這個序列與其他醣蛋白(具有各種生理作用的醣蛋白胎球蛋白和作為生物藥品使用的促紅血球生成素)的一端連接,就會產生Lewis X修飾。
此外,研究團隊還利用鄰近依賴性標記法(為目標蛋白質附近存在的分子做標記的方法)實驗發現,FUT9附近存在很多連接這29個胺基酸殘基的促紅血球生成素。這表明,在Lewis X獨特性修飾中,29個胺基酸殘基的序列可能具有在細胞中促進與FUT9會合的作用。
加藤教授表示:「今後,通過研究各種醣蛋白有望發現新的分子密碼。通過將其作為糖鏈修飾的控制碼使用,希望能為開發出更多的生物藥品以及更高端的細胞醫療做出貢獻」。
原文:《科學新聞》
翻譯編輯:JST客觀日本編輯部
【論文資訊】
期刊:Communications Biology
論文:An embeddable molecular code for Lewis X modification through interaction with fucosyltransferase 9
DOI:10.1038/s42003-022-03616-1