理化學研究所(理研)BDR中心(Center for Biosystems Dynamics Research)的金堅石博士, 小川泰策博士(參與研究時),北條望博士和城口克之課題組長(教授)以及東京大學定量生命科學研究所岡崎拓教授、清水謙次助教,東海大學醫學部基礎醫學系今西規教授、Kirill Kryukov奨勵研究員(參與研究時),東京理科大學生命醫科學研究所伊川友活教授等組成的研究團隊成功開發出了一套基於活細胞圖像推斷全轉錄組定義細胞分子表型[1]的新平台。該平台通過對同一單細胞分別測定活細胞影片圖像和全轉錄組,結合深度學習,成功從活細胞動態圖像直接推斷出了全轉錄組定義的細胞分子表型。

理化學研究所BDR中心(Center for Biosystems Dynamics Research)的金堅石博士在此次開發的 「ALPS」前。(供圖:理研)
由於能夠快速、低成本地鑑定包括細胞類型和細胞狀態在内的細胞分子表型,所以該研究的應用前景廣闊,比如可以應用於細胞治療中植入前評估細胞的狀態。由於不再需要使用鑑定細胞類型的分子標記試劑而節省成本。此外,對分子標籤未知或者很難製備分子標記試劑的細胞類型或狀態也是非常有效的策略。
研究團隊此次開發出了一套可自動進行顯微鏡成像和單細胞分選的機器人「ALPS(Automated Live-imaging and Cell Picking System)」。ALPS可自動化影片成像培養皿中一定區域内的每個細胞,通過逐一分離到96孔PCR板中,可在大約16分鐘内成像和分離96個細胞。在以三種血液細胞為對象的實驗中,研究團隊利用二代測序技術 [2]對ALPS成像並分離的細胞進行了單細胞RNA測序分析[3],合計測定了1,000多個細胞的全轉錄組;同時研究團隊還使用人工智慧的深度學習[4]技術,提取出了細胞圖像與全轉錄組之間的關係,從中推斷出了細胞的全轉錄組定義的類型或狀態。相關内容已於2022年12月28日發布在科學雜誌《Proceedings of the National Academy of Sciences of the United States of America(PNAS)》線上版上。
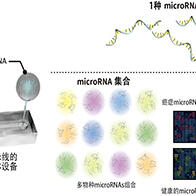
圖1 從單細胞圖像推斷全轉錄組定義的細胞類型或狀態(供圖:理研)
近年來,人們發現雖然構成組織和細胞群的細胞看起來很相似,但實際上有許多不同的類型和狀態。所以在細胞研究中,重要的是確定細胞的類型和狀態。確定細胞的類型和狀態的典型方法是單細胞RNA測序技術,用以全面測量細胞中表達的RNA總類和數量,從而測定每個細胞的全轉錄組狀態。然而,這一方法一般需要將細胞破碎後再進行分析,因此很難測量動態變化的狀態,比如測量同一個單細胞在不同時間的全轉錄組。
作為一種非侵入式識別細胞類型和狀態的方法,基於圖像的人工智慧(深度學習)方法正在引起人們的關注。人工智慧可以通過學習大量被標記了狀態(如癌症等)的細胞或組織圖像來獲得圖像與細胞狀態之間的關係,從而對新的細胞圖像推斷出細胞的狀態。
「ALPS」實現細胞圖像高速採集和全轉錄成分析
此次研究團隊開發的自動化細胞成像和分選的機器人「ALPS」,實現了對同一細胞的影片圖像高速採集和全轉錄成分析(圖2)。ALPS可通過光學顯微鏡自動重複觀察大量細胞,並可通過即時分析圖像來檢測細胞特性,最後根據細胞的圖像結果自動化分選細胞。同時為了對分選後的細胞進行全轉錄組測序,研究團隊還對ALPS分選得到的96個細胞同時進行自動化測序建庫。在細胞分選中,細胞的吸取和放入PCR管中的步驟都通過相機即時記錄和確認。該系統還可用於絕對數少的細胞和細胞集團的純化。
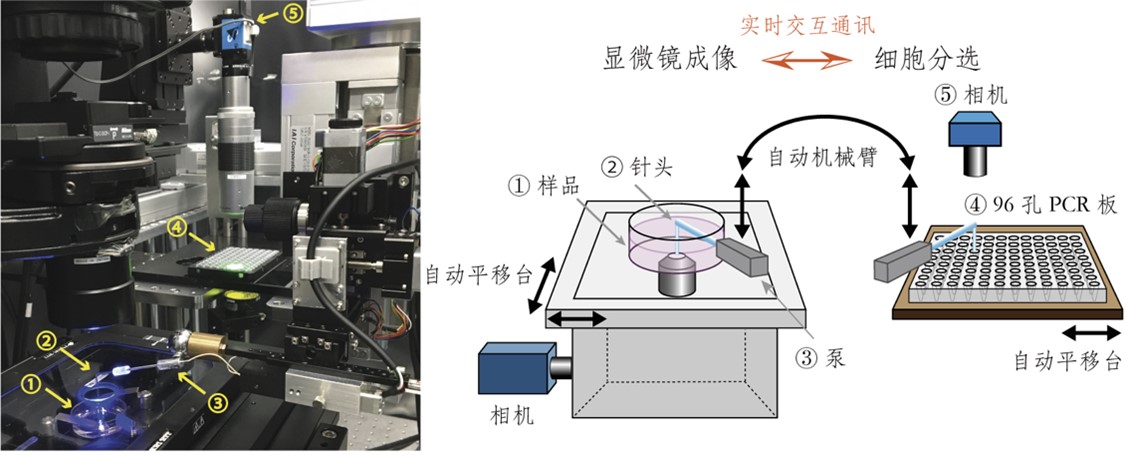
圖2自動細胞成像與分選機器人「ALPS」 (供圖:理研)
研究團隊使用ALPS和自動化測序建庫系統實際分析了混合有多種小鼠血液系統培養細胞的樣本,並獲得了總計1,000多個細胞的時間序列活細胞圖像和全轉錄組數據集。基於每個單細胞的全轉錄組數據集,這些細胞被鑑定為三種細胞類型(T細胞、白血病細胞和造血祖細胞)(圖3)。
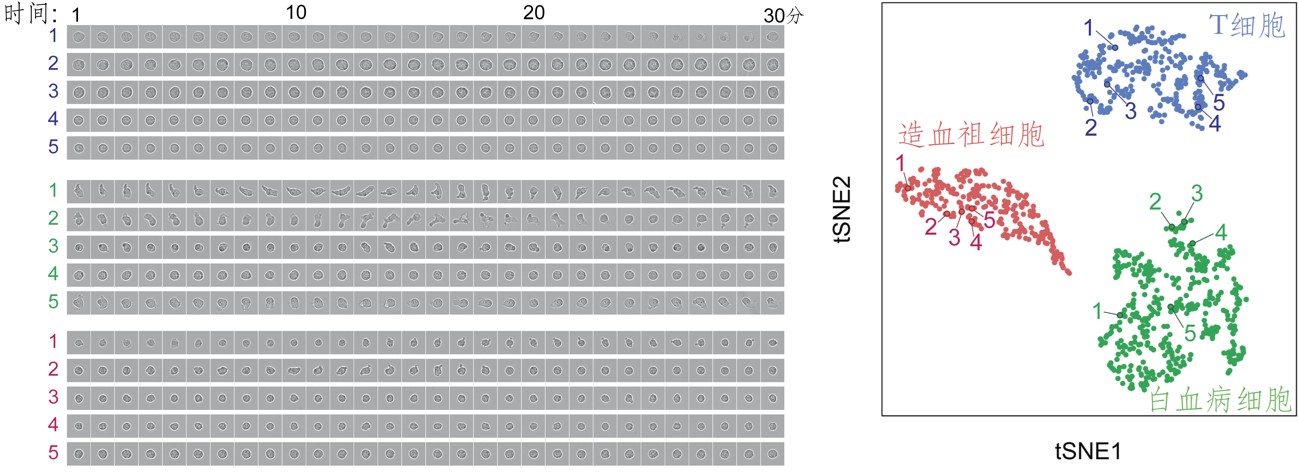
圖3同一細胞的圖像與轉錄組數據集(供圖:理研)
上圖左為T細胞(藍色)、白血病細胞(綠色)和造血祖細胞(紅色)隨時間變化的活細胞圖像(每分鐘拍攝一幀,持續拍攝30分鐘)。上圖右為由ALPS成像後分離得到細胞的轉錄成分析結果。該分析採用了t相鄰域嵌入方法(tSNE法),在二維圖中投影每個細胞全轉錄組的相對區別,圖中一個點代表一個細胞。通過聚類分析全轉錄組,細胞被分成了三組,每組代表一種細胞類型。左右圖中數字表示相同的細胞,所有細胞都獲得了圖像和轉錄組數據。
使用該數據集,研究團隊嘗試了從時間序列活細胞圖像中直接推斷轉錄組鑑定的細胞類型。首先,使用數據集中大約95%的細胞,對深度學習模型進行訓練來提取圖像和轉錄組定義細胞類型之間的相關。然後,使用深度學習獲得的模型從時間序列活細胞圖像中推測剩餘5%細胞的細胞類型。研究團隊比較了深度學習推測的細胞類型以及通過轉錄組鑑定的細胞類型,發現推測的準確率達到了81%(圖4)。由此研究團隊認為深度學習有能力從細胞圖像中直接推斷出由轉錄組定義的三種細胞類型(T細胞、白血病細胞和造血祖細胞)。
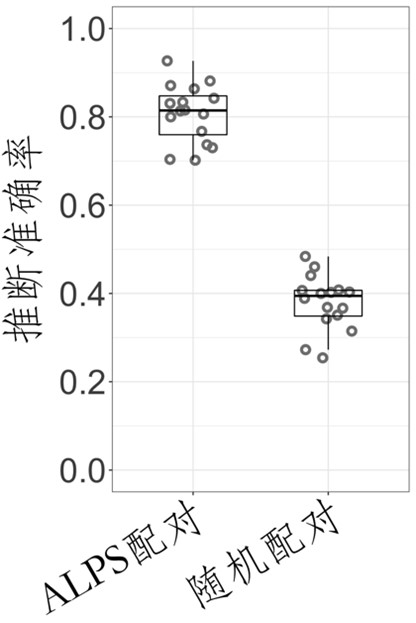
圖4從細胞圖像推斷細胞類型時的準確率(供圖:理研)
從時間序列活細胞圖像推斷三種由全轉錄組定義的細胞類型時,其推斷準確率可達81%。「ALPS配對」是指用APLS正確連接的細胞圖像和全轉錄組定義的細胞類型的數據集來進行的深度學習結果。「隨機配對」是指用隨機標記了細胞類型的細胞圖像數據集來進行的深度學習結果。「ALPS配對」和「隨機配對」的明顯區別表明,深度學習可以提取細胞圖像和轉錄組定義細胞類型之間的相關。
為了測試該方法是否可以區分同一細胞類型的不同細胞狀態時,研究團隊首先通過轉錄組鑑定出了造血祖細胞中具有不同基因表現模式的兩種細胞狀態,然後驗證了深度學習可以通過時間序列活細胞圖像區分這兩種細胞狀態。另外,為了證明這種方法對從活生物體中直接獲取的細胞也有效,研究團隊用該方法直接分析了小鼠外周血單核球(PBMC)。結果顯示,深度學習能夠從時間序列活細胞圖像顯著區分由轉錄組鑑定的三種核心免疫細胞,即B細胞、CD4+T細胞和CD8+T細胞。這些結果都表明,深度學習可以從細胞圖像直接推斷出多種全轉錄組確定的細胞類型和狀態。
本次研究開發的基於活細胞圖像推斷全轉錄組定義細胞分子表型的技術平台可以說是近年來備受關注的BioDX[5] 的成果之一,有望進一步推廣BioDX並為各種生命科學研究做出貢獻。本研究由理化學研究所運營基金(BDR中心「DECODE項目」)和文部科學省創新領域科學研基金(研究領域提案)資助(課題負責人:城口克之)。
名詞解釋
[1]全轉錄組定義的細胞分子表型
即使具有相同DNA序列的細胞也可以通過表達不同的基因來執行不同的功能。在本文中,以被測量到的所有基因的表達量來定義的細胞分子表型被稱為「全轉錄組定義的細胞分子表型」。
[2]二代測序技術
與「一代測序技術」相對的術語,「一代測序技術」現在主要指Sanger測序法。「二代測序技術」相比「一代測序技術」,其測序通量提高了幾個數規模,一次可以確定大約107到1010(1000萬到100億)個DNA序列。
[3]單細胞RNA測序技術
利用高通量DNA測序儀分析單個細胞中所含RNA(主要是互補DNA)的序列,從而全面定量確定基因的種類和數量的方法。
[4]人工智慧(AI)(深度學習)
深度學習是人工智慧(AI)的一種方法,用於訓練計算機以學習的方式處理數據。深度學習模型可以識別圖像和其他數據中的複雜模式,從而實現準確的預測。
[5]BioDX
生命科學研究中的「數字化轉型(DX)」。預計利用資訊科技實現創新飛躍。
文 曹暉 JST客觀日本
【論文資訊】
雜誌:Proceedings of the National Academy of Sciences of the United States of America(PNAS)
論文:Robotic data acquisition with deep learning enables cell image–based prediction of transcriptomic phenotypes
DOI:doi.org/10.1073/pnas.2210283120
【相關研究】
理研等成功開發出高通量菌群分析新技術,開闢單細胞水平菌群測定的先河
理研開發出精確測量腸道菌數量和種類的方法,可掌握微妙的變化