日本國立遺傳學研究所發生遺傳學研究室川上浩一教授的研究團隊與大阪羽曳野醫療中心森秀夫醫師等人發現了一種無法通過常規PCR檢查判斷陽性的變異株。相關成果已發表在《Cureus》上。
為了診斷是否感染COVID-19(新冠肺炎)的致病病毒SARS-CoV2(新型冠狀病毒),一般採用RT-qPCR(逆轉錄定量聚合酶鏈式反應)法進行檢查。在RT-qPCR法中,在SARS-CoV2的N基因的2個區域設定的引子和探針序列被日本寶生物(Takara Bio)、東洋紡(TOYOBO)等公司製造的RT-qPCR試劑盒所利用。
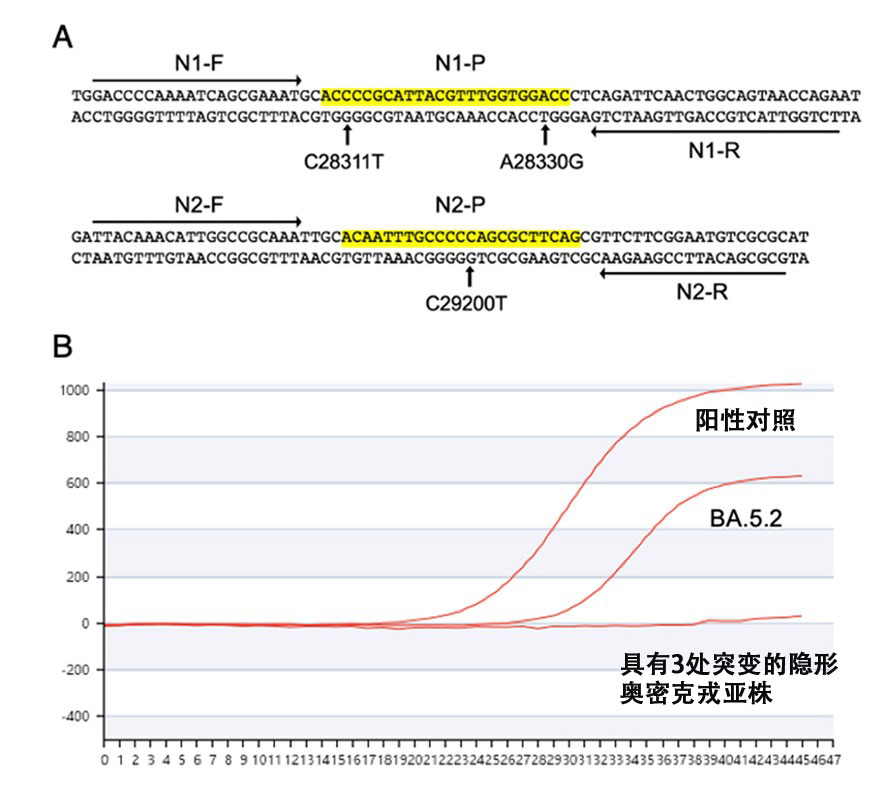
圖:(A)N1和N2區域的引子和探針序列以及本次發現的變異位置。箭頭(N1-F、N1-R、N2-F、N2-R)表示引子的位置。標黃區域表示探針(N1-P和N2-P)的位置。在患者樣本中發現了C28311T、A28330G和C29200T的變異。
(B)使用N1和N2引子探針進行的RT-qPCR擴增曲線。
使用寶生物公司製造RT-qPCR試劑盒分析陽性對照、在N1區域有2個突變的BA.5.2,以及在N1區域有2個突變和在N2區域有1個突變的變異體的結果。(供圖:國立遺傳學研究所)
新冠疫情大流行以來,SARS-CoV2出現了多種基因體突變的亞株,目前奧密克戎亞株是世界和日本的主要流行毒株。
去年10月中旬,在大阪一名發燒患者身上採集到了一份口水樣本,儘管通過多種不同的核酸檢測被診斷為陽性,但使用N1和N2引子探針的RT-qPCR試劑盒卻文能檢測出SARS-CoV2。研究團隊於是對這份不可思議的樣本進行了詳細分析。
對口水樣本的S基因(3)區域的分析表明,這個感染株為奧密克戎的一個亞系變種。研究團隊猜想該亞系變種的N基因可能存在未知突變,通過詳細檢查N1和N2區域的鹼基序列,發現N1區域有2處(C28311T和A28330G)、N2區域有1處(C29200T) 突變。這些突變被認為是使用引子的傳統RT-qPCR檢測試劑盒無法正常發揮作用的原因。另一個已知的奧密克戎變種BA5.2在N1區域有2處突變(C28311T和A28330G),但在N2區域沒有突變。
此次用寶生物公司生產的RT-qPCR試劑盒對樣本進行分析後發現,對於BA5.2,PCR擴增產物雖然為陽性對照的一半左右,但還可以檢測到,而該樣本並未因實施PCR而擴增產物出現增加。
在N1和N2區域有3處突變的突變體,在日本及世界所有病毒序列中分別隻發現了0.23%和0.06%。考慮到某些檢測試劑盒無法檢測到具有這3處突變的病毒,因此這一頻率極有可能低於實際情況。換言之,在使用N1和N2引子探針的PCR檢查中,有可能漏掉了一部分COVID-19患者。
所以,如果有COVID-19的特徵性症狀,在使用常規引子的PCR檢測中無法確定為陽性時,則需要通過在不同位置使用引子和探針進行PCR檢測,以避免遺漏這種變異株。
原文:《科學新聞》
翻譯:JST客觀日本編輯部
【論文資訊】
雜誌:Cureus
論文:Stealth Omicron: a novel SARS-CoV-2 variant that is insensitive to RT-qPCR using the N1 and N2 primer-probes
DOI:10.7759/cureus.36373