東京大學生產技術研究所坪山幸太郎講師(研究當時為美國西北大學博士後)和美國西北大學Gabriel Rocklin助教等人組成的研究團隊成功構建了一種高效測量蛋白質結構穩定性的方法。蛋白質的結構穩定性是衡量蛋白質保持特定功能結構的指標,並定義了功能蛋白質分子的比例。以往的方法一次實驗只能測量一種蛋白質的結構穩定性,但此次開發的新方法,一次實驗可以測量約90萬種蛋白質的結構穩定性。相關研究成果已發表在《Nature》上。
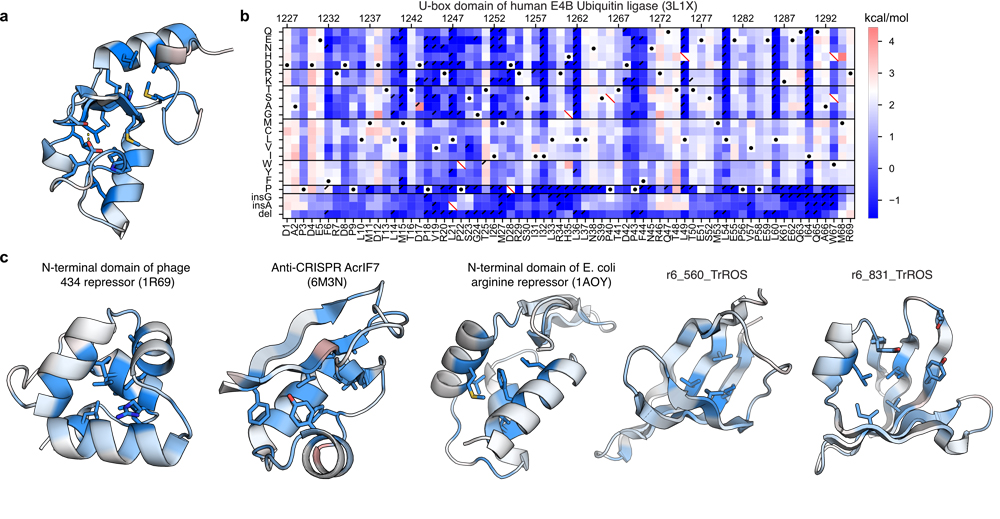
圖1 蛋白質結構穩定性的解析圖(供圖:東京大學)
(a,c)顯示了每個蛋白質位置對結構穩定性的重要性(藍色越深越重要,紅色越深越不重要)。(b)(a)蛋白質結構圖的原始數據。與野生型蛋白質的結構穩定性(白、黑點)相比,結構穩定性更低(藍)或結構穩定性更高(紅)。為了製作單個蛋白質的「解剖圖」,需要測量千種以上的胺基酸序列的結構穩定性。橫軸代表蛋白質中的位置,縱軸代表氨基酸的置換、缺失和插入。
關於生物體內的蛋白質,人們熟知的是「胺基酸序列決定了蛋白質的結構,進而決定其功能」的安芬森法則。幾乎所有蛋白質的功能都由其結構決定。此外,大多數蛋白質會在無結構的解體狀態和整齊摺疊的結構狀態等多個狀態之間轉換。因此,顯示蛋白質整齊摺疊結構的分子比例的「蛋白質的結構穩定性」,即代表了功能性蛋白質的比例,是蛋白質最重要的特徵數據之一。
事實上,當蛋白質的結構穩定性降低時,可能導致蛋白質之間發生非預期的相輔作用,或者導致功能性蛋白質不足,從而引發癌症等各種疾病。
儘管如此重要,但正確理解蛋白質的結構穩定性,根據蛋白質的胺基酸序列和結構來預測蛋白質的結構穩定性是很困難的,需要耗費大量的心力、成本和時間。因此,即使整合了過去數十年來測定的蛋白質結構穩定性的數據庫,也不過隻包含了約3萬種蛋白質的資訊。此外,由於這些資訊是從各種研究論文中整合而來,測定條件和資料品質各異,因此難以綜合理解蛋白質的結構穩定性。
研究團隊通過在兩個方面下功夫,開發了一種高效測量蛋白質結構穩定性的方法。
第一個方面是,將蛋白質的胺基酸序列資訊轉換成DNA核酸資訊。利用可以將蛋白質與相應的cDNA相結合的cDNA顯示法,擷取cDNA序列來確定胺基酸序列。通過結合cDNA顯示法和次世代DNA測序技術兩種技術,實現了一次解析大量類型的蛋白質胺基酸序列。
第二個方面是,為了量化蛋白質的結構穩定性,使用了蛋白質切割酶。蛋白酶可以切割已經解體的不安定穩態的蛋白質,結構穩定性較高的蛋白質可以被緩慢地切斷,而結構穩定性較低的蛋白質會被迅速切割。也就是說,通過測量蛋白質被切割的速度,可以測量蛋白質的結構穩定性。
由此研究團隊成功地在一次實驗中測量了約90萬種蛋白質的結構穩定性。研究人員多次重複該實驗,從中選擇高質量的數據,最終獲得並公開了約80萬種蛋白質結構穩定性的數據組。該數據庫不僅規模龐大,而且具備在同一測量條件下測量的優勢。
此外,研究人員還研究了約500種天然蛋白質和人工蛋白質,調查了各個蛋白質結構的保持機制。針對每個蛋白質,分別對每個位置的氨基酸進行了20種不同的氨基酸置換和缺失,並插入甘胺酸或丙胺酸測量了其結構穩定性。以此實現了對每個蛋白質中哪些部位對維持結構穩定性至關重要等結構穩定性的視覺化。
同時,研究人員還調查了生物體內蛋白質中各種氨基酸的易用性與氨基酸帶來的結構穩定性之間的關係。發現越是能穩定結構的氨基酸越容易被使用,並且能夠量化這種趨勢。此外,還研究了排除結構穩定性的影響時氨基酸的使用情況,結果顯示,易溶於水的親水性氨基酸更容易被使用,而不易溶於水的疏水性氨基酸,尤其是含有芳香環的芳香族氨基酸,使用頻率較低。
由於該數據庫與以往相比規模龐大,因此有可能揭示蛋白質結構穩定性的重要性質以及與結構穩定性相關的普遍規律。此外,大規模的結構穩定性數據將成為蛋白質科學AI開發的基礎。有望通過這種AI幫助識別導致疾病的氨基酸突變或更有效地合成蛋白質藥物。
原文:《科學新聞》
翻譯:JST客觀日本編輯部
【論文資訊】
雜誌:Nature
論文:Mega-scale experimental analysis of protein folding stability in biology and design
DOI:10.1038/s41586-023-06328-6