靜岡縣立大學的中野祥吾副教授和碩士1年級學生小澤洋樹的研究團隊發表研究成果稱,開發了一種通過在計算機中虛擬演化蛋白質來設計高功能蛋白質的新方法「GAOptimizer法」。實驗證實,將該方法應用於結構、序列和功能各不相同的3種蛋白質後,設計出的人工蛋白質與作為模板的天然來源蛋白質相比,具有更適合利用的優秀功能。此次的成果有望實現天然來源蛋白質的高功能化設計,並加速開發使用蛋白質的下一代生物材料。相關成果已刊登在《Cell Reports Physical Science》上。
另外,GAOptimizer可通過網頁(https://zenodo.org/records/10208126)下載。
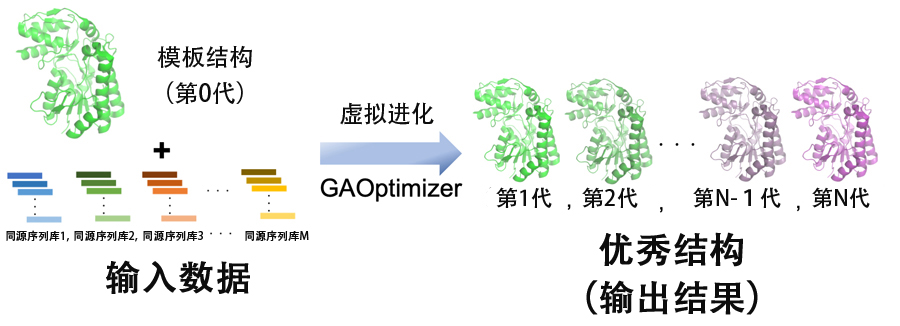
圖1.GAOptimizer概要圖。GAOptimizer把需要高功能化的蛋白質三維結構(模板結構)及其同源性物1)蛋白質胺基酸序列作為輸入資料,進行N代虛擬演化。演化後每一代的優秀結構將作為輸出結果被保存下來。N的數量可由客戶自由設定。(供圖:靜岡縣立大學)
研究團隊把需要改善功能的目標蛋白(模板結構)及其同源性序列作為輸入資料,找出改善功能的有用突變點,並利用基因演算法開發出了最適化多點有用突變的組合以引入目標蛋白質的新蛋白質設計方法「GAOptimizer法」。
研究人員首先從30個父代結構中引入交叉重組突變(從父代結構中隨機選擇2個結構並將其交叉)或隨機突變(從父代結構中選擇1個結構並引入點突變),生成100個雜交世代結構。其次,使用適應度函數計算出生成的子結構的得分數。適應度函數使用評估蛋白質結構穩定性的Rosetta評分(REU),以及其他不依賴於結構穩定性的評分。根據計算出的分值,選擇適應度最好的子結構作為優秀結構,並保存為第n代的代表結構。此外,從生成的100個雜交世代結構中隨機選擇5個結構,並選出其中適應度最好的子結構作為父代結構的候選結構。通過重複這一過程30次,從子結構中選出下一代(n+1代)的30個父代結構,使其完成一代的演化。重複這個計算N代,完成虛擬演化。
通過將適應度函數更改為REU以外的分數而虛擬演化得到的S-羥基腈裂解酶(S-HNL)與天然型HNL相比,生產量提高10倍以上、催化效率提高3倍以上、耐熱性(Tm)提高約5度,更靠適合使用。
原文:《科學新聞》
翻譯:JST客觀日本編輯部
【論文資訊】
雜誌:Cell Reports Physical Science
論文:Development of Evolutionary Algorithm-Based Protein Redesign Method
DOI:10.1016/j.xcrp.2023.101758