為深入理解複雜的生態系統,必須精確掌握生物種類及其豐度。對於肉眼可見的大型生物,通常能夠通過直接觀察進行計數。然而,對於大小僅為幾微米且數量龐大的細菌,採用「計數DNA(基因)」的方法才是有效的。理化學研究所生命機能科學研究中心(理研BDR)細胞系統動態預測研究團隊的負責人城口克之等人在《iMetaOmics》期刊上發表綜述文章,概括了該技術領域的發展歷程,通過介紹標記基因解析由多種細菌構成的「細菌群集」生態系統的技術,闡述了該技術對未來生命科學研究的潛在影響。
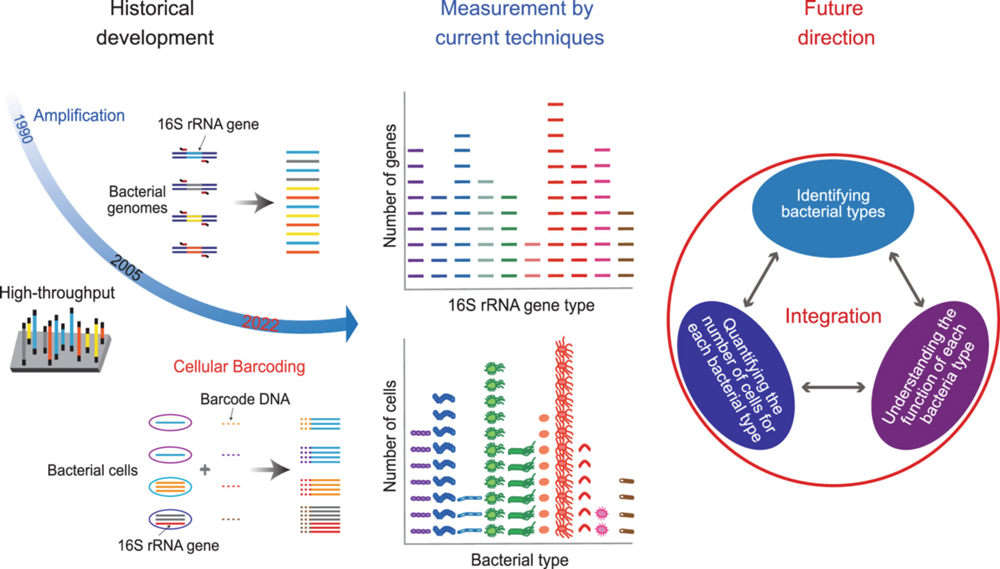
基於16S rRNA基因進行細菌物種鑑定和細胞數定量,並結合對各細菌功能的理解進行綜合的細菌群集分析。(供圖:理化學研究所生命機能科學研究中心、原圖出自iMetaOmics刊登論文)
用於細菌群集解析的主要標記基因是16S核糖體RNA(16S rRNA)基因。這一區域的DNA序列在不同物種之間一般具有顯著差異,因此成為鑑定細菌群集中存在細菌的重要線索。自1977年該方法首次被論證以來,已經經過了多次開發與改進。現在,這一分析方法已成為研究與人類健康密切相關的共生細菌群集的重要工具。
城口克之團隊負責人等在2022年開發了一種新型細菌群集測量方法——「BarBIQ(Barcoding Bacteria for Identification and Quantification)法」注)。這種方法雖然與傳統方法一樣使用16S rRNA基因測序,但能夠更準確地區分細菌,並且可以精確測量細菌的細胞數量。使用BarBIQ法,可以準確捕捉諸如飲食習慣變化引起的腸道細菌群集的微小變化。
關於細菌群集研究的未來發展,城口克之團隊負責人指出:「目前還存在無法區分非常近緣的細菌種類的情況,所以還有進一步改進的空間」。然而,即便在現階段,利用現有技術獲取定量和定性數據仍具有重要意義。通過BarBIQ法,人類現在能夠準確瞭解多種細菌的細胞數量和存在比例。如果將這些資訊與每種細菌的全基因體解析所得到的基因和代謝通路等功能資訊相結合,就能夠更深入地理解整個細菌群集的行為。此外,通過整合基於細胞數測量的細菌分泌物分子量等其他數據,並進行數學分析,有可能準確預測因干預等因素引起的細菌群集變化。
生命科學的發展一直依賴於分析技術的進步以及對生命現象的理解。城口克之團隊負責人在其綜述中總結指出:「在細菌群集研究中,新的測量技術帶來的變革期即將到來」。
文 理化學研究所生命機能科學研究中心 宣傳部
注)參見2022年2月22日 客觀日本 「理研等成功開發出高通量菌群分析新技術,開闢單細胞水平菌群測定的先河」
【論文資訊】
期刊:iMetaOmics
論文:Long journey of 16S rRNA-amplicon sequencing toward cell-based
functional bacterial microbiota characterization.
DOI: doi.org/10.1002/imo2.9